세포는 우리 몸을 구성하는 가장 기본 단위로 사람의 성장, 발달 및 노화뿐만 아니라 다양한 질병의 발생 및 진행을 일으키는 기본 단위이다. 우리 몸은 약 35조개 이상의 세포로 구성되며, 이들 세포는 신경, 상피, 근육, 면역, 섬유아 세포 등을 포함하여 약 220종류의 세포로 분류된다. 여러 종류의 세포들은 조직을 이루고, 이들 조직은 장기를 구성하며, 장기들은 모여서 인체를 구성한다. 인체에 생기는 질병은 특정 장기의 조직에서 발생하며, 이는 해당 조직을 구성하는 세포들의 이상이 근본적인 원인이다.
예를 들어, 위암의 경우 위의 선상 조직(glandular tissue)에서 상피세포의 비정상적인 세포분열에 의해 발생하며, 또한 조직 내의 섬유아 및 면역 세포들은 세포 간 상호작용을 통해 암 상피세포에 영향을 줌으로써 암의 다양한 표현형(진행, 전이 및 약물 반응성 등)을 결정한다. 하지만 같은 섬유아, 면역세포라도 암 가까이 있는 세포들과 멀리 있는 세포들은 다른 기능적 특성을 가진다. 암 조직 내의 각 상피 세포들도 가지고 있는 유전자 변이 및 세포 간 상호작용이 달라서 서로 다른 특성을 가질 수 있다.
인간세포지도 프로젝트,
RNA 시퀀싱에서 시작되다
이런 세포들 간 이질성(heterogeneity)은 암의 표현형을 이해하는 데 중요하다. 예를 들면, 전이성이 큰 위암 조직은 전이성이 없는 위암 조직에 비해 조직 내에 전이를 유도하는 기능을 가진 섬유아 세포가 존재할 수 있고, 또 전이를 억제하는 기능을 가진 특정 면역세포가 없을 수 있다. 특정 조직을 구성하는 세포들의 이질성은 기능적으로 분류된 세포의 종류, 각 세포 종류별 비율, 각 세포 종류의 공간적 분포 등으로 정의된다. 기능적으로 세포의 분류는 세포가 가지는 유전자 변이 상태, mRNA, 단백질 발현 패턴 및 대사 작용 패턴 등으로 정의된다.
최근 오믹스 기술의 발달로 단일세포 레벨에서 유전자 변이를 측정하는 단일세포 게놈 시퀀싱(single cell whole genome sequencing), mRNA의 발현량을 측정하는 단일세포 RNA 시퀀싱(single cell RNA sequencing), 단백질의 발현량을 측정하는 단일세포 단백체 분석(single cell proteomics analysis)이 가능하게 되었다. 기존에는 조직을 통째로 오믹스 분석을 하기 때문에, 다양한 세포 종류들의 정보들이 섞여 측정됨으로써, 암을 분류할 때(전이암과 비전이암, 약물 민감성과 저항성을 보이는 암 등), 유전자 변이나 mRNA, 단백질의 발현량의 차이가 어떤 세포에서 유래한 것인지 구분할 수 없어서, 세포 이질성에 의해 정의되는 암의 표현형을 제대로 규명할 수 없다는 한계점이 지적되어 왔다.
상기된 단일세포 오믹스 분석기술 중 가장 보편적으로 적용되는 기술은 단일세포 RNA 시퀀싱이다. 미국, 영국, 독일, 싱가포르, 한국 등 세계 여러 나라의 연구자들이 참여하는 인간세포지도(HCA·Human Cell Atlas) 공동연구 프로젝트에서는 다양한 주요 장기(뇌, 신장, 폐, 간, 췌장, 위, 대장, 심장, 피부, 면역 시스템 등)에 대해서 공간적으로 어떤 세포들이 어떻게 장기를 구성하는가를 보여주는 단일세포 레벨에서의 세포 지도를 작성하는 것을 목표로 하고 있다.
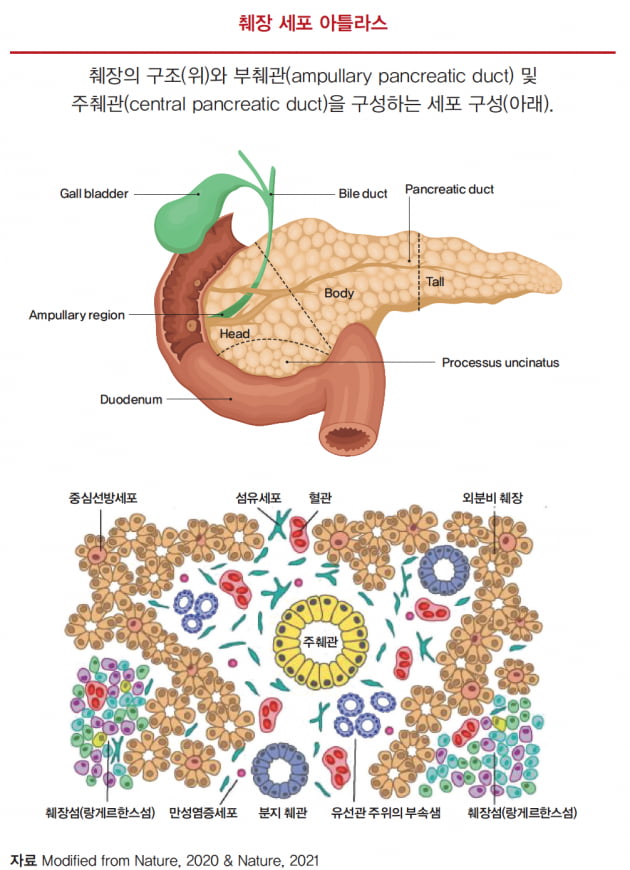
예를 들면, 혈당을 조절하는 인슐린·글루카곤 호르몬을 분비하고 소화효소를 분비해 음식물을 소화시키는 기능을 하는 췌장(오른쪽 위)에 대해, 공간 단일세포 RNA 시퀀싱(spatial single cell RNA sequencing) 분석을 통해 세포지도가 구성되고 있다.
오른쪽의 아래 그림은 부췌관·주췌관을 구성하는 단일세포 레벨의 세포지도를 보여주고 있다. 이러한 세포지도는 각 세포에서 mRNA 발현 패턴 및 세포들의 공간적 분포를 바탕으로 예측된 세포간 상호작용을 제공한다. 이들 정보에 기반하여 어떤 세포들이 어떻게 상호작용하면서 췌장의 기능인 호르몬 및 소화효소 분비를 하는가를 설명할 수 있다.
단백질 발현 패턴은 어떻게 변하는가
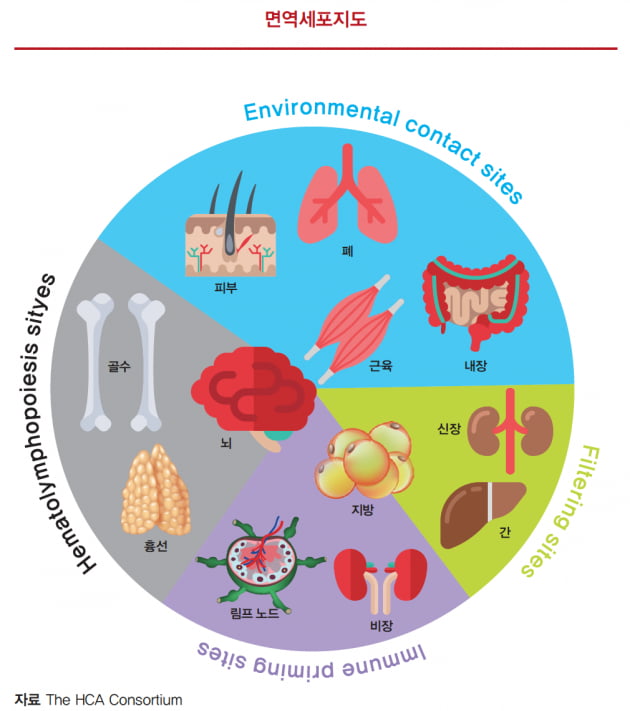
다른 장기들과는 다르게 면역 시스템을 구성하는 면역세포들은 특정 장기에 국한되어 존재하기보단 다양한 장기에 존재하며 외부 병원체로부터 해당 장기를 보호한다.
면역세포들이 존재하는 사이트들은 크게 외부 환경과 접촉(environmental contact site), 필터링(filtering site), 면역 활성(immune priming site), 혈액·임파구 생성 사이트(hematolymphopoiesis site)로 나뉜다. 면역세포지도는 각 사이트로 분류된 장기·조직에 존재하는 면역세포들의 종류(B·T 세포, 대식세포, 호중성·호산성·단핵 백혈구, 또는 이들의 서브 세포군), 세포 종류별 비율, 각 세포에서의 mRNA 발현 패턴 등의 정보를 제공한다.
이들 정보에 기반하여 해당 조직, 장기에서의 서로 다른 면역반응을 단일세포 레벨에서 설명하는 것이 가능해진다. 예를 들면, 흉선에서는 항원 특이적으로 적응면역반응(adaptive immune response)을 일으키는 T 세포 성숙되는 장소이다. 이들 T 세포들을 분리하여 단일세포 게놈, RNA 시퀀싱으로 분석하면, 특정 항원에 대해 면역 메모리를 가지는 T세포의 특성을 규명할 수 있다. 이러한 관점에서 코로나 감염에서 회복한 환자의 흉선에서 뽑은 T세포들에 대한 단일세포 분석을 통해 많은 제약사 및 연구자들이 백신을 개발하려는 시도를 하고 있다.
인간세포지도 프로젝트의 1단계에서 구축된 다양한 장기 및 면역 시스템에 대한 세포지도들은 정상 상태에서 세포들이 어떻게 상호작용하면서 해당 장기의 기능을 하는가에 대한 근본적인 정보를 제공한다. 인간세포지도 프로젝트의 2단계에서는 정상 장기 세포지도들이 각 장기에서 질병이 발병, 진행하면서 단일세포 레벨에서 각 구성 세포들이 어떻게 유전자 변이나 mRNA, 단백질 발현량 패턴이 변하는 정보를 기반으로 정상 세포지도의 질병 상태로의 이행을 이해하는 것을 목표로 한다.
예를 들어, 췌장암 환자의 췌장에서 암이 발병하고 진행 및 재발하는 동안 상기된 췌장 및 면역세포지도의 통합적 이행을 이해하면, 어떤 세포군에서 어떤 세포경로에 속하는 유전자의 mRNA 발현량의 증가가 암의 발병, 전이, 재발에 중요한 역할을 하는지 규명할 수 있다.
이들 정보를 기반으로 증가된 세포경로에 속하는 주요 단백질의 기능을 억제하는 약물을 디자인하여 치료 효과가 개선된 새로운 췌장암 치료제를 개발할 수도 있고, 또는 현재 사용되는 약물 중에서 치료 효과가 좋은 최적의 치료제를 선별할 수 있다. 같은 방법을 이용해 인간세포지도는 주요 장기에서 발병하는 난치성 질환의 치료법 개발에 큰 공헌을 할 것으로 기대하고 있다.
현재는 단일세포 레벨의 mRNA 발현량에 의해 인간세포지도가 구축되고 있다. 인간세포지도의 3단계 프로젝트는 다중 단일세포 분석(single cell multiomics)을 통해 각 세포에 대해 mRNA뿐만 아니라 유전자 변이 및 단백질 발현량 정보까지 제공하는 다층세포지도(multilayered cell atlas) 구축과 이를 통해 질병에 따른 다층세포지도의 이행을 이해하는 것을 목표로 한다. 단일세포 레벨의 유전자 변이를 측정하는 단일세포 게놈 시퀀싱 기술은 지속적으로 발전하고 있어, 현재보다 정확한 각 세포의 유전자 변이 정보를 제공할 수 있을 것으로 기대된다.
현재는 시퀀싱 에러와 단일염기변이(single nucleotide polymorphism)의 구분이 명확하지 않으나, 기술의 발달로 유전자 변이 정보가 정확해지면 암세포의 클론진화(clonal evolution) 분석을 통해 전이성 암세포 또는 약물 저항성 암세포들의 기원이 되는 세포의 정체와 특징을 규명할 수 있고, 이들을 사멸할 수 있는 약물을 디자인하여 암의 근간을 치료하는 것이 가능해질 수 있다. 중합효소연쇄반응(PCR·Polymerase Chain Reaction)에 의해 증폭이 가능한 유전자 및 mRNA와 달리, 단백질은 증폭이 불가능하다.
따라서 수천, 수만개 유전자의 변이, mRNA의 발현양 측정할 수 있는 단일세포 게놈, mRNA 시퀀싱에 비해, 단일세포 단백체 분석은 약 100개 이하의 단백질에 대해 발현량을 측정할 수 있는 한계점이 있다. 다양한 기술이 개발, 적용되고 있으나 근본적으로 단일세포에 존재하는 단백질의 양이 작아서 민감도가 획기적으로 큰 기술이 개발되기 전까지는 측정 가능한 단일세포 단백체(single cell proteome) 사이즈는 느리게 증가할 것으로 기대된다.
마지막으로 다층세포지도 및 질병 진행에 따른 다층세포지도의 이행을 효율적으로 분석하여 각 장기의 기능에 있어 주요 세포, 이들 세포에서 주요 세포경로 정보 및 질병 치료를 위한 타깃 분자(유전자·mRNA·단백질 등)를 선별하는 생명정보학적 방법론이 매우 중요하다. 기본적으로 단일세포 오믹스 분석은 각 세포에서 존재하는 분자의 양이 적기 때문에 측정값은 심한 노이즈에 의해 변질되거나 측정값이 없는 분자들이 많이 존재하게 된다. 이러한 노이즈와 데이터의 희박성으로 인해 장기의 기능 또는 질병의 표현형을 결정하는 주요 세포군, 세포군의 기능을 정의하는 세포경로 및 활성을 조절할 수 있는 주요 분자 타깃을 신뢰성 있게 선별할 수 있는 정보학적 방법론의 개발이 활발하게 진행되고 있다.
또 다층세포지도가 제공하는 유전자 변이, mRNA, 단백질의 발현량은 각 정보가 가지는 파워가 다르다. mRNA 발현량은 수천 유전자에 대해 존재하지만, 질병에 관련된 체세포 변이(somatic mutation)는 소수의 유전자에서 나타나므로 빈도가 낮고, 단백질 발현량은 약 100개의 단백질에 대해서만 측정값이 존재한다. 따라서 mRNA 정보가 체세포 변이나 단백질 발현량에 비해 큰 데이터 파워를 가지게 된다. 이런 상황에서 데이터 파워가 틀린 정보들을 효율적으로 분석하여 모든 데이터가 일관되게 지시하는 세포경로 및 주요 분자 타깃을 동정하는 정보학적 방법론 개발은 매우 중요하다.
조직을 통째로 분석하여 생산된 다중 오믹스 데이터는 단일세포 데이터에 비해 노이즈와 데이터의 희박성이 상대적으로 적다.
이들 조직 다중 오믹스 데이터의 분석을 위해 개발된 방법론들의 적용 및 다양한 변경이 시도되고 있으나, 단일세포 다중 오믹스 데이터의 특이적인 노이즈와 데이터 희박성의 문제를 만족스럽게 해결하지는 못하고 있어, 앞으로 새로운 방법론 개발이 절실하게 요구되고 있다.
결론적으로 인간세포지도 프로젝트의 3단계까지 진행되어 인체의 주요 장기에 대한 다층세포지도 및 질병 진행 동안 다층세포지도의 이행이 규명되고, 이들 단일세포 정보를 효율적으로 분석할 수 있는 생명정보학적 방법론이 개발된다면, 현재 조직 레벨 데이터로부터 얻을 수 있는 이해의 한계점을 극복함으로써 난치성 질환의 치료에 있어서 단일세포 레벨의 정보에 기반한 새로운 치료법들을 개발할 수 있을 것이다.
<저자 소개>

서울대 생명공학부 교수로 재직 중이다. 포스텍에서 학사와 석사를 마친 뒤 미국 매사추세츠공대(MIT)에서 박사 학위를 취득했다. 이후 포스텍, DGIST 교수를 거쳐 서울대에서 생물정보학을 연구하고 있다. 지난해 3월, 조기발병 위암의 유전단백체를 분석한 공로로 과학기술정보통신부와 한국연구재단이 선정하는 이달의 과학기술인에 선정됐다. 한국생명공학연구원의 공통기반기술 관련 연구과제의 자문을 맡은 바 있다.
*이 글은 <한경바이오인사이트> 매거진 2021년 4월호에 실렸습니다.